数据预处理
其中一种需要删除的变量是常数自变量,或者是方差极小的自变量,对应的命令是nearZeroVar。
zerovar=nearZeroVar(mdrrDescr)
newdata1=mdrrDescr[,-zerovar]
另一类需要删除的是与其它自变量有很强相关性的变量,对应的命令是findcorrelation。自变量中还有可能存在多重共线性问题,可以用findLinearCombos命令将它们找出来。
descrCorr = cor(newdata1)
highCorr = findCorrelation(descrCorr, 0.90)
newdata2 = newdata1[, -highCorr]
comboInfo = findLinearCombos(newdata2)
newdata2=newdata2[, -comboInfo$remove]
我们还需要将数据进行标准化并补足缺失值,这时可以用preProcess命令,缺省参数是标准化数据,其高级功能还包括用K近邻和装袋决策树两种方法来预测缺失值。此外它还可以进行cox幂变换和主成分提取。
Process = preProcess(newdata2)
newdata3 = predict(Process, newdata2)
最后是用createDataPartition将数据进行划分,分成75%的训练样本和25%检验样本,类似的命令还包括了createResample用来进行简单的自助法抽样,还有createFolds来生成多重交叉检验样本。
inTrain = createDataPartition(mdrrClass, p = 3/4, list = FALSE)
trainx = newdata3[inTrain,]
testx = newdata3[-inTrain,]
trainy = mdrrClass[inTrain]
testy = mdrrClass[-inTrain]
在建模前还可以对样本数据进行图形观察
featurePlot(trainx[,1:2],trainy,plot='box')

特征选择
首先定义几个整数,程序必须测试这些数目的自变量
subsets = c(20,30,40,50,60,70,80)
然后定义控制参数,functions是确定用什么样的模型进行自变量排序,本例选择的模型是随机森林即rfFuncs,可以选择的还有lmFuncs(线性回归),nbFuncs(朴素贝叶斯),treebagFuncs(装袋决策树),caretFuncs(自定义的训练模型)。method是确定用什么样的抽样方法,本例使用cv即交叉检验, 还有提升boot以及留一交叉检验LOOCV。
ctrl= rfeControl(functions = rfFuncs, method = "cv",verbose = FALSE, returnResamp = "final")
最后使用rfe命令进行特征选择,计算量很大,这得花点时间
Profile = rfe(newdata3, mdrrClass, sizes = subsets, rfeControl = ctrl)
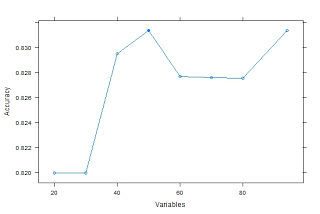
用图形发现当选取50个自变量的时候精度最好
plot(Profile)
下面的命令则可以返回最终保留的自变量
Profile$optVariables
建模与参数优化
首先得到经过特征选择后的样本数据,并划分为训练样本和检验样本
newdata4=newdata3[,Profile$optVariables]
inTrain = createDataPartition(mdrrClass, p = 3/4, list = FALSE)
trainx = newdata4[inTrain,]
testx = newdata4[-inTrain,]
trainy = mdrrClass[inTrain]
testy = mdrrClass[-inTrain]
然后定义模型训练参数,method确定多次交叉检验的抽样方法,number确定了划分的重数, repeats确定了反复次数。
fitControl = trainControl(method = "repeatedcv", number = 10, repeats = 3,returnResamp = "all")
确定参数选择范围,本例建模准备使用gbm算法,相应的参数有如下三项
gbmGrid = expand.grid(.interaction.depth = c(1, 3),.n.trees = c(50, 100, 150, 200, 250, 300),.shrinkage = 0.1)
利用train函数进行训练,使用的建模方法为提升决策树方法,
gbmFit1 = train(trainx,trainy,method = "gbm",trControl = fitControl,tuneGrid = gbmGrid,verbose = FALSE)
用图形观察结果,interaction.depth取1,n.trees取150时精度最高

另一种得到预测结果的方法是使用extractPrediction函数
predValues = extractPrediction(models,testX = testx, testY = testy)
head(predValues)
从中可提取检验样本的预测结果
testValues = subset(predValues, dataType == "Test")
如果要得到预测概率,则使用extractProb函数
probValues = extractProb(models,testX = testx, testY = testy)
testProbs = subset(probValues, dataType == "Test")
对于分类问题的效能检验,最重要的是观察预测结果的混淆矩阵
Pred1 = subset(testValues, model == "gbm")
Pred2 = subset(testValues, model == "treebag")
confusionMatrix(Pred1$pred, Pred1$obs)
confusionMatrix(Pred2$pred, Pred2$obs)
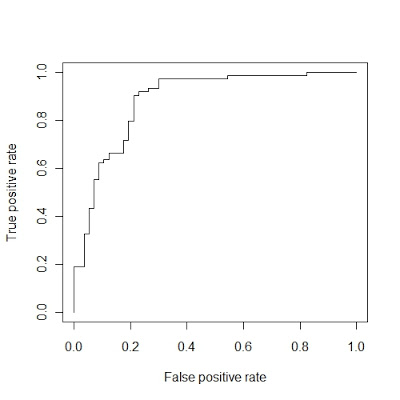
最后是利用ROCR包来绘制ROC图
prob1 = subset(testProbs, model == "gbm")
prob2 = subset(testProbs, model == "treebag")
library(ROCR)
prob1$lable=ifelse(prob1$obs=='Active',yes=1,0)
pred1 = prediction(prob1$Active,prob1$lable)
perf1 = performance(pred1, measure="tpr", x.measure="fpr" )
plot( perf1 )